-
Notifications
You must be signed in to change notification settings - Fork 1
/
Copy pathexperimentalDesignI.Rmd
650 lines (467 loc) · 22.3 KB
/
experimentalDesignI.Rmd
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
124
125
126
127
128
129
130
131
132
133
134
135
136
137
138
139
140
141
142
143
144
145
146
147
148
149
150
151
152
153
154
155
156
157
158
159
160
161
162
163
164
165
166
167
168
169
170
171
172
173
174
175
176
177
178
179
180
181
182
183
184
185
186
187
188
189
190
191
192
193
194
195
196
197
198
199
200
201
202
203
204
205
206
207
208
209
210
211
212
213
214
215
216
217
218
219
220
221
222
223
224
225
226
227
228
229
230
231
232
233
234
235
236
237
238
239
240
241
242
243
244
245
246
247
248
249
250
251
252
253
254
255
256
257
258
259
260
261
262
263
264
265
266
267
268
269
270
271
272
273
274
275
276
277
278
279
280
281
282
283
284
285
286
287
288
289
290
291
292
293
294
295
296
297
298
299
300
301
302
303
304
305
306
307
308
309
310
311
312
313
314
315
316
317
318
319
320
321
322
323
324
325
326
327
328
329
330
331
332
333
334
335
336
337
338
339
340
341
342
343
344
345
346
347
348
349
350
351
352
353
354
355
356
357
358
359
360
361
362
363
364
365
366
367
368
369
370
371
372
373
374
375
376
377
378
379
380
381
382
383
384
385
386
387
388
389
390
391
392
393
394
395
396
397
398
399
400
401
402
403
404
405
406
407
408
409
410
411
412
413
414
415
416
417
418
419
420
421
422
423
424
425
426
427
428
429
430
431
432
433
434
435
436
437
438
439
440
441
442
443
444
445
446
447
448
449
450
451
452
453
454
455
456
457
458
459
460
461
462
463
464
465
466
467
468
469
470
471
472
473
474
475
476
477
478
479
480
481
482
483
484
485
486
487
488
489
490
491
492
493
494
495
496
497
498
499
500
501
502
503
504
505
506
507
508
509
510
511
512
513
514
515
516
517
518
519
520
521
522
523
524
525
526
527
528
529
530
531
532
533
534
535
536
537
538
539
540
541
542
543
544
545
546
547
548
549
550
551
552
553
554
555
556
557
558
559
560
561
562
563
564
565
566
567
568
569
570
571
572
573
574
575
576
577
578
579
580
581
582
583
584
585
586
587
588
589
590
591
592
593
594
595
596
597
598
599
600
601
602
603
604
605
606
607
608
609
610
611
612
613
614
615
616
617
618
619
620
621
622
623
624
625
626
627
628
629
630
631
632
633
634
635
636
637
638
639
640
641
642
643
644
645
646
647
648
649
650
---
title: "Experimenteel Design I: replicatie en power"
author: "Lieven Clement"
date: "statOmics, Ghent University (https://statomics.github.io)"
output:
html_document:
code_download: yes
theme: cosmo
toc: yes
toc_float: yes
highlight: tango
number_sections: yes
---
<a rel="license" href="https://creativecommons.org/licenses/by-nc-sa/4.0"><img alt="Creative Commons License" style="border-width:0" src="https://i.creativecommons.org/l/by-nc-sa/4.0/88x31.png" /></a>
```{r setup, include=FALSE}
knitr::opts_chunk$set(include = TRUE, comment = NA, echo = TRUE,
message = FALSE, warning = FALSE)
library(tidyverse)
```
```{r pop2Samp2Pop,fig.asp=.8, fig.align='center',echo=FALSE}
if ("pi"%in%ls()) rm("pi")
kopvoeter<-function(x,y,angle=0,l=.2,cex.dot=.5,pch=19,col="black")
{
angle=angle/180*pi
points(x,y,cex=cex.dot,pch=pch,col=col)
lines(c(x,x+l*cos(-pi/2+angle)),c(y,y+l*sin(-pi/2+angle)),col=col)
lines(c(x+l/2*cos(-pi/2+angle),x+l/2*cos(-pi/2+angle)+l/4*cos(angle)),c(y+l/2*sin(-pi/2+angle),y+l/2*sin(-pi/2+angle)+l/4*sin(angle)),col=col)
lines(c(x+l/2*cos(-pi/2+angle),x+l/2*cos(-pi/2+angle)+l/4*cos(pi+angle)),c(y+l/2*sin(-pi/2+angle),y+l/2*sin(-pi/2+angle)+l/4*sin(pi+angle)),col=col)
lines(c(x+l*cos(-pi/2+angle),x+l*cos(-pi/2+angle)+l/2*cos(-pi/2+pi/4+angle)),c(y+l*sin(-pi/2+angle),y+l*sin(-pi/2+angle)+l/2*sin(-pi/2+pi/4+angle)),col=col)
lines(c(x+l*cos(-pi/2+angle),x+l*cos(-pi/2+angle)+l/2*cos(-pi/2-pi/4+angle)),c(y+l*sin(-pi/2+angle),y+l*sin(-pi/2+angle)+l/2*sin(-pi/2-pi/4+angle)),col=col)
}
par(mar=c(0,0,0,0),mai=c(0,0,0,0))
plot(0,0,xlab="",ylab="",xlim=c(0,10),ylim=c(0,10),col=0,xaxt="none",yaxt="none",axes=FALSE)
rect(0,6,10,10,border="red",lwd=2)
text(.5,8,"populatie",srt=90,col="red",cex=2)
symbols (3, 8, circles=1.5, col="red",add=TRUE,fg="red",inches=FALSE,lwd=2)
grid=seq(0,1.3,.01)
for (i in 1:50)
{
angle1=runif(n=1,min=0,max=360)
angle2=runif(n=1,min=0,max=360)
radius=sample(grid,prob=grid^2*pi/sum(grid^2*pi),size=1)
kopvoeter(3+radius*cos(angle1/180*pi),8+radius*sin(angle1/180*pi),angle=angle2)
}
text(7.5,8,"Effect van roken in populatie",col="red",cex=1.2)
rect(0,0,10,4,border="blue",lwd=2)
text(.5,2,"sample",srt=90,col="blue",cex=2)
symbols (3, 2, circles=1.5, col="red",add=TRUE,fg="blue",inches=FALSE,lwd=2)
for (i in 0:2)
for (j in 0:4)
{
kopvoeter(2.1+j*(3.9-2.1)/4,1.1+i)
}
text(7.5,2,"Effect van roken in steekproef",col="blue",cex=1.2)
arrows(3,5.9,3,4.1,col="black",lwd=3)
arrows(7,4.1,7,5.9,col="black",lwd=3)
text(1.5,5,"EXP. DESIGN (1)",col="black",cex=1.2)
text(8.5,5,"SCHATTEN &\nINFERENTIE (3)",col="black",cex=1.2)
text(7.5,.5,"DATA EXPLORATIE &\nBESCHRIJVENDE STATISTIEK (2)",col="black",cex=1.2)
```
# Concepten
- Experimentele eenheden zijn subjecten, planten, proefdieren of objecten die aan behandelingen worden onderworpen in een experimentele studie.
- Observationele eenheden zijn de subjecten, planten proefdieren of objecten waarop we metingen doen. Meestal zijn de experimentele en observationele eenheden hetzelfde. Als dat niet het geval is heeft met vaak pseudoreplicatie.
- Experimentele eenheden representatief voor populatie: Randomisatie
- Replicatie: technisch vs biologisch, steekproefgrootte - power
- Bronnen van variabiliteit: technisch, biologisch, binnen en tussen subjecten.
# Replicatie
Paper over replicatie in Nature Methods (2 pagina's) [[PDF](https://www.nature.com/articles/nmeth.3091.pdf)]
{ width=100% }
## Voorbeeld
In een RNA-seq experiment willen de onderzoekers het effect nagaan van een medicijn op de gen expressie.
Mogelijkse onderzoeksvraag
- Is er een verschil in gemiddelde gen expressie tussen behandelde en niet behandelde samples
## Bronnen van variabiliteit
TABLE 1 NATURE METHODS | VOL.11 NO.9 | SEPTEMBER 2014 | 879 - 880
| | Replicate type | Replicate category$^\text{a}$ |
|:---:|:---|:---:|
| | Colonies | B |
| | Strains | B |
| Animal study subjects | Cohoused groups | B |
| | Gender | B |
| | Individuals | B |
| | | |
| | Organs from sacrificed animals | B |
| | Methods for dissociating cells from tissue | T |
| Sample preparation | Dissociation runs from given tissue sample | T |
| | Individual cells | B |
| | | |
| | RNA-seq library construction | T |
| | Runs from the library of a given cell | T |
| Sequencing | Reads from different transcript molecules | V$^\text{b}$ |
| | Reads with unique molecular identifier (UMI) from a given transcript molecule | T |
(a) Replicates are categorized as biological (B), technical (T) or of variable type (V).
(b) Sequence reads serve diverse purposes depending on the application and how reads are used in analysis.
---
## Op welk niveau moeten we repliceren?
- $\text{var}(X)=\sigma^2_A+\sigma^2_C+\sigma^2_M=\sigma^2_{TOT}$
- $\text{var}(\bar{X})=\frac{\sigma^2_A}{n_A}+\frac{\sigma^2_C}{n_A n_C} + \frac{\sigma^2_M}{n_A n_C n_M}$
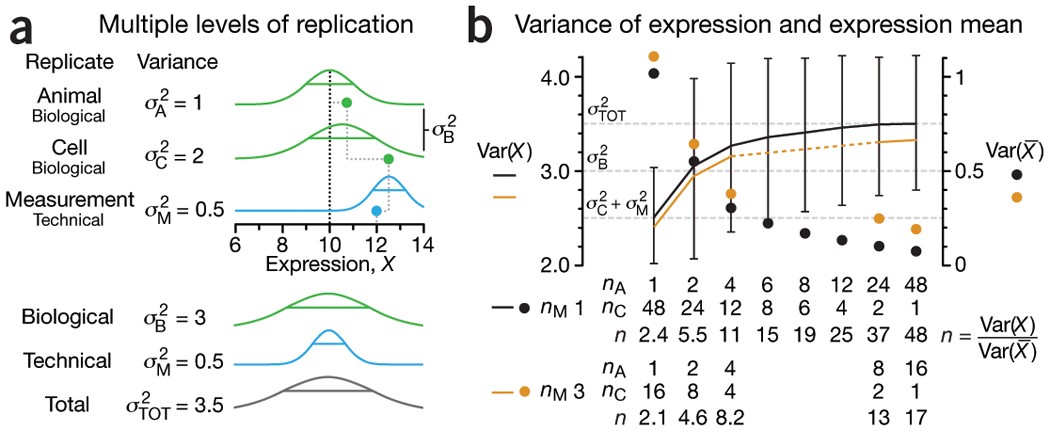
Figure 1 NATURE METHODS | VOL.11 NO.9 | SEPTEMBER 2014 | 879 - 880
(a) Three levels of replication (two biological, one technical) with animal, cell and measurement replicates normally distributed with a mean across animals of 10 and ratio of variances 1:2:0.5. Solid green (biological) and blue (technical) dots show how a measurement of the expression (X = 12) samples from all three sources of variation. Distribution s.d. is shown as horizontal lines.
(b) Expression variance, Var(X), and variance of expression mean, Var($\bar X$), computed across 10,000 simulations of nAnCnM = 48 measurements for unique combinations of the number of animals (nA = 1 to 48), cells per animal (nC = 1 to 48) and technical replicate measurements per cell (nM = 1 and 3). The ratio of Var(X) and Var($\bar X$) is the effective sample size, n, which corresponds to the equivalent number of statistically independent measurements. Horizontal dashed lines correspond to biological and total variation. Error bars on Var(X) show s.d. from the 10,000 simulated samples (nM = 1).
---
Waar ligt interesse?
- Karakteriseren van bronnen van variabiliteit in experiment ==> technische en biologische repeats zijn nodig
- Voornamelijke interesse in schatten van effect van behandeling ==> focus op biologische repeats
- Extra biologische repeats lumpen alle bronnen van variabiliteit: biologische + technische variabiliteit.
- Goed experimenteel design vereist na denken over replicatie
1. Identificeer onderzoeksvragen die men wenst te beantwoorden.
2. Breng bronnen van variabiliteit in kaart in elke stap van het experiment
3. Let op voor pseudoreplicatie, beoog zoveel mogelijk onafhankelijke repeats.
---
# Power, steekproefgrootte en andere design aspecten.
Reading materials:
[Nature Methods (2013), 10(12), 1139–1140](https://www.nature.com/articles/nmeth.2738.pdf)
## Intermezzo lineare regression in matrix vorm
- Lineaire regressie is een belangrijke methode in statistische data analyse.
### Scalaire vorm
- Stel dat men voor elke experimentele eenheid $p$ predictoren meet $\mathbf{x}=(x_1,\ldots,x_p)$ en
- continue respons $Y$
- het linear regressiemodel kan dan worden geschreven als:
\[
Y=f(\mathbf{x}) +\epsilon=\beta_0+\sum\limits_{j=1}^p x_j\beta_j + \epsilon
\]
met i.i.d. $\epsilon\sim N(0,\sigma^2)$
### Vector/Matrix vorm
- $n$ observatie $(\mathbf{x}_1,y_1) \ldots (\mathbf{x}_n,y_n)$ with $\mathbf{x}_1^T=[1 x_1 \ldots x_p]$
- Regressie in matrix notatie
\[\mathbf{Y}=\mathbf{X\beta} + \mathbf{\epsilon}\]
met $\mathbf{Y}=\left[\begin{array}{c}y_1\\ \vdots\\y_n\end{array}\right]$,
$\mathbf{X}=\left[\begin{array}{cccc} 1&x_{11}&\ldots&x_{1p}\\
\vdots&\vdots&&\vdots\\
1&x_{n1}&\ldots&x_{np}
\end{array}\right]$ or $\mathbf{X}=\left[\begin{array}{c} \mathbf{x}_1^T\\\vdots\\\mathbf{x}_n^T\end{array}\right]$,
$\boldsymbol{\beta}=\left[\begin{array}{c}\beta_0\\ \vdots\\ \beta_p\end{array}\right]$ and
$\mathbf{\epsilon}=\left[\begin{array}{c} \epsilon_1 \\ \vdots \\ \epsilon_n\end{array}\right]$
---
## kleinste kwadraten techniek/Least Squares (LS)
- Minimimaliseer de residuele kwadratensom
\begin{eqnarray*}
RSS(\boldsymbol{\beta})&=&\sum\limits_{i=1}^n e^2_i\\
&=&\sum\limits_{i=1}^n \left(y_i-\beta_0-\sum\limits_{j=1}^p x_{ij}\beta_j\right)^2
\end{eqnarray*}
- of in matrix notatie
$$
\text{RSS}(\boldsymbol{\beta})=(\mathbf{Y}-\mathbf{X\beta})^T(\mathbf{Y}-\mathbf{X\beta})
$$
$$\rightarrow \hat{\boldsymbol{\beta}}=\text{argmin}_\beta \text{ RSS}(\boldsymbol{\beta})$$
---
### Minimimaliseer RSS
\[
\begin{array}{ccc}
\frac{\partial RSS}{\partial \boldsymbol{\beta}}&=&\mathbf{0}\\\\
\frac{(\mathbf{Y}-\mathbf{X\beta})^T(\mathbf{Y}-\mathbf{X}\boldsymbol{\beta})}{\partial \boldsymbol{\beta}}&=&\mathbf{0}\\\\
-2\mathbf{X}^T(\mathbf{Y}-\mathbf{X}\boldsymbol{\beta})&=&\mathbf{0}\\\\
\mathbf{X}^T\mathbf{X\beta}&=&\mathbf{X}^T\mathbf{Y}\\\\
\hat{\boldsymbol{\beta}}&=&(\mathbf{X}^T\mathbf{X})^{-1}\mathbf{X}^T\mathbf{Y}
\end{array}
\]
---
## Variantie schatter?
\[
\begin{array}{ccl}
\hat{\boldsymbol{\Sigma}}_{\hat{\boldsymbol{\beta}}}
&=&\text{var}\left[(\mathbf{X}^T\mathbf{X})^{-1}\mathbf{X}^T\mathbf{Y}\right]\\\\
&=&(\mathbf{X}^T\mathbf{X})^{-1}\mathbf{X}^T\text{var}\left[\mathbf{Y}\right]\mathbf{X}(\mathbf{X}^T\mathbf{X})^{-1}\\\\
&=&(\mathbf{X}^T\mathbf{X})^{-1}\mathbf{X}^T(\mathbf{I}\sigma^2)\mathbf{X}(\mathbf{X}^T\mathbf{X})^{-1}
\\\\
&=&(\mathbf{X}^T\mathbf{X})^{-1}\mathbf{X}^T\mathbf{I}\quad\mathbf{X}(\mathbf{X}^T\mathbf{X})^{-1}\sigma^2\\\\
%\hat{\boldmath{\Sigma}}_{\hat{\boldsymbol{\beta}}}&=&(\mathbf{X}^T\mathbf{X})^{-1}\mathbf{X}^T\text{var}\left[\mathbf{Y}\right](\mathbf{X}^T\mathbf{X})^{-1}\mathbf{X}\\
&=&(\mathbf{X}^T\mathbf{X})^{-1}\mathbf{X}^T\mathbf{X}(\mathbf{X}^T\mathbf{X})^{-1}\sigma^2\\\\
&=&(\mathbf{X}^T\mathbf{X})^{-1}\sigma^2
\end{array}
\]
De onzekerheid op model parameters hangt dus af van de residuele variabiliteit en het de proefopzet!
- Hoe groter $\mathbf{X}^T\mathbf{X}$ hoe meer informatie het experiment zal hebben over de model parameters en hoe kleiner hun variantie en standaard errors!
- Factoriële designs?
- Designs with continue predictoren?
---
De effectgrootte van interesse is typisch een lineaire combinatie van de model parameters,
$$
l_0 \times \beta_0 + l_1 \times \beta_1 + ... + l_{p-1} \times \beta_{p-1} = \mathbf{L}^T\boldsymbol{\beta}
$$
De nulhypothese van onze test kan dan worden geschreven als
$$
H_0: \mathbf{L}^T\boldsymbol{\beta} = 0
$$
vs de alternatieve hypothese
$$
H_1: \mathbf{L}^T\boldsymbol{\beta} \neq 0
$$
En het bewijs in het experiment tegen $H_0$ kan worden gekwantificeerd met de t-test statistiek:
$$
t=\frac{\mathbf{L}^T\hat{\boldsymbol{\beta}} - 0}{\text{se}_{\mathbf{L}^T\hat{\boldsymbol{\beta}}}}
$$
die een t-distributie volgt met n-p vrijheidsgraden onder de nul hypothese als alle aannames van het model geldig zijn.
De kracht van de toets is dan
$$P(p < 0.05 | H_1)$$
en hangt af van
- de werkelijke effectgrootte in de populatie $\mathbf{L}^T\boldsymbol{\beta}$.
- Het aantal observaties: SE en df van t-test.
- Keuze van de designpunten
- Keuze van significantie-niveau $\alpha$.
We kunnen dit evalueren aan de hand van simulaties.
## Muis voorbeeld
- In 2021 publiceerden Choa et al. dat cytokine Thymic stromal lymphopoietin
(TSLP) vetverlies induceert bij muizen die een vet dieet krijgen door de secretie van talg. [[html](https://www.science.org/doi/full/10.1126/science.abd2893)] [[PDF](https://www.science.org/doi/pdf/10.1126/science.abd2893)]
- Stel dat je een gelijkaardig experiment op wil zetten om na te gaan of cytokine interleukin 25 (IL) ook een effect heeft op het gewicht van muizen.
- Je plant een studie met een controle groep muizen die een hoog vet dieet krijgen (HFD) en een behandelingsgroep die een HFD dieet met IL krijgt.
- Wat is de vereiste steekproefgrootte om een bepaald effect op te pikken van de behandeling?
### Hoe zal je de data van het experiment analyseren?
---
- $H_0$: Er is geen verschil in het gewicht van muizen die een dieet met IL krijgen en muizen die het controle dieet krijgen.
- $H_1$: Het gewicht is gemiddelde verschillend tussen muizen die het IL dieet krijgen en muizen die het controle dieet krijgen.
- Two sample t-test of een t-test op de helling van het lineaire model met 1 dummy variabele
$$ Y_i = \beta_0 + \beta_1 X_\text{iIL} + \epsilon_i$$
$$
\text{met }
X_\text{iIL}=\begin{cases}X_{iIL}=0 & \text{HFD}\\X_{iIL}=1 & \text{HFD + IL}\end{cases}.
$$
- Geschatte effectgrootte?
$$\hat\delta = \bar X_{IL} - \bar X_{c} = \hat \beta_1$$
- Test statistiek
$$
T = \frac{\bar{X}_{IL}-\bar{X}_{c}}{SD_\text{pooled} \times \sqrt{\frac{1}{n_1} + \frac{1}{n_2}}} = \frac{\hat\beta_1}{\text{SE}_{\hat\beta_1}}
$$
- $\hat \beta_1$ is een onvertekende schatter van het werkelijk gewichtsverschil ($\delta =\mu_{IL}-\mu_{c} = \beta_1$) dat zou voorkomen in de populatie van muizen gevoed met HFD vs muizen gevoed met HFD + IL.
### Power?
$$
P[p < \alpha \vert \beta_1 \neq 0]
$$
De kracht (power) zal dus afhangen van
- Het werkelijke gemiddelde gewichtsverschil $\beta_1$ in de populatie.
- De variabiliteit van de gewichtsmetingen
- het significantie-niveau $\alpha$
- steekproefgrootte $n_{IL}$ en $n_c$ in beide groepen
We kunnen de power schatten als
1. voldaan is aan de aannamens van het model: gewicht in beide groepen is normaal verdeeld met gelijke variantie
en als we de
2. standaard deviatie kennen van gewichtsmetingen rond hun groepsgemiddelde voor muizen die een HFD diet krijgen
3. de echte effectgrootte in de populatie
4. $n_1$ en $n_2$
### Gebruik data uit vorig experiment om inzicht te krijgen in de gewichtsdata van muizen
Stel dat we toegang hebben tot data van een vorig experiment (hier dat van Karen Svenson en Dan Gatti, gefinancierd door P50 GM070683 en beschikbaar op [PH525x](http://genomicsclass.github.io/book/pages/random_variables.html))
```{r}
mice <- read.csv("https://raw.githubusercontent.com/genomicsclass/dagdata/master/inst/extdata/femaleMiceWeights.csv")
mice %>%
ggplot(aes(x=Diet,y=Bodyweight)) +
geom_boxplot(outlier.shape=FALSE) +
geom_jitter()
mice %>%
ggplot(aes(sample=Bodyweight)) +
geom_qq() +
geom_qq_line() +
facet_wrap(~Diet)
```
```{r}
mice <- mice %>% mutate(Diet = as.factor(Diet))
miceSum <- mice %>%
group_by(Diet) %>%
summarize_at("Bodyweight",list(mean=~mean(.,na.rm=TRUE),
sd=~sd(.,na.rm=TRUE),
n=function(x) x%>%is.na%>%`!`%>%sum)) %>%
mutate(se=sd/sqrt(n))
miceSum
```
In het experiment werden twee diëten gebruikt:
- gewoon diet o.b.v. granen (Chow)
- Heel vet dieet (high fat: hf)
We kunnen de data van de hf muizen gebruiken als input voor onze power analyse.
- De data van hf muizen lijken normaal verdeeld te zijn.
- Het gemiddelde gewicht van een hf muis is `r miceSum %>% filter(Diet == "hf") %>% pull("mean") %>% round(.,1)`g
- De standaard deviatie (SD) van het gewicht van hf muizen is `r miceSum %>% filter(Diet == "hf") %>% pull("sd") %>% round(.,1)`g
Effectgroote?
- De alternative hypothese is complex.
- Omvat alle mogelijke effecten!
- Om een power analyse te doen kiezen we daarom een minimale effectgrootte die we wensen op te pikken in het nieuwe experiment.
- Veronderstel dat we een gewichtsverschil willen oppikken van minimaal 10%.
```{r}
delta <- - round(miceSum$mean[2] *.1,1)
delta
```
Merk op dat het gemiddeld gewicht dan dicht komt bij dat van muizen in het pilootexperiment die werden gevoed met het normale dieet.
We kunnen nu een simulatie studie opzetten om de power te berekenen voor een experiment met 8 muizen in elke groep.
#### Eén simulatie
```{r}
set.seed(1423)
n1 <- n2 <- 8
b0 <- round(miceSum$mean[2],1)
b1 <- - delta
sd <- round(miceSum$sd[2],1)
alpha <- 0.05
x <- rep(0:1,c(n1,n2))
y <- b0 + b1 * x + rnorm(n1+n2,0, sd = sd)
fit <- lm(y~x)
bhat <- fit$coef
stat <- summary(fit)
summary(fit)$coef[2,]
```
Voor het gesimuleerde experiment konden we het effect van de behandeling `r ifelse(summary(fit)$coef[2,"Pr(>|t|)"] < alpha,"","niet")` oppikken bij het $\alpha$=`r alpha` (p = `r round(summary(fit)$coef[2,"Pr(>|t|)"],2)`) significantie-niveau!
- We moeten het experiment herhalen om de power te schatten.
- We zullen de simulatie daarom coderen in een functie die we kunnen hergebruiken.
#### Simulatie van herhaalde experimenten (repeated experiments)
```{r}
library(multcomp)
n1 <- n2 <- 8
b0 <- round(miceSum$mean[2],1)
b1 <- - delta
sd <- round(miceSum$sd[2],1)
predictorData <- data.frame(Diet = rep(c("c","hf"),c(n1,n2)) %>% as.factor)
alpha <- 0.05
simLm <- function(form, data, betas, sd, contrasts, simIndex = NA)
{
dataSim <- data
X <- model.matrix(form,dataSim)
dataSim$ySim <- X%*%betas + rnorm(nrow(dataSim),0,sd)
form <- formula(paste("ySim ~",form[[2]]))
fitSim <- lm(form,dataSim)
mcp <- glht(fitSim,linfct = contrasts)
return(summary(mcp)$test[c("coefficients","sigma","tstat","pvalues")]%>% unlist)
}
simLm(
form = ~Diet,
data = predictorData,
betas = c(b0,b1),
sd = sd,
contrasts = "Diethf = 0")
```
We hebben nu een generiek functie waarmee we data kunnen simuleren die normaal verdeeld zijn voor elk mogelijk design die we met het lineair model kunnen analyseren.
De functie heeft volgende argumenten:
- `form`: One sided formula including the structure of the predictors in the model
- `data`: Data frame with the predictor values for the design
- `betas`: A vector with values for all mean model parameters
- `sd`: The standard deviation of the error
- `contrasts`: a scalar or a vector with the null hypotheses that we would like to assess.
- `simIndex`: an arbitrary argument that is not used by the function but that will allow it to be used in an sapply loop that runs from 1 up to the number of simulations.
Simuleer nSim = 1000 herhaalde experimenten:
```{r}
set.seed(1425)
nSim <- 1000
simResults <- t(sapply(1:nSim,simLm,form = ~Diet,data = predictorData,betas = c(b0,b1),sd = sd,contrasts = "Diethf = 0"))
power <- mean(simResults[,4] < alpha)
power
```
We hebben een power van `r power*100`% om het behandelingseffect op te pikken met 8 biologische herhalingen in elke behandelingsgroep.
Merk op dat de power van een experiment ook het hoogste is als het aantal observaties gebalanceerd is, m.a.w. $n_1=n_2$.
#### Power voor verschillende steekproefgroottes
We kunnen de power nu berekenen voor meerdere steekproefgrootes.
```{r}
power <- data.frame(n=c(3,5,10,25,50,75,100),power=NA)
for (i in 1:nrow(power))
{
n1 <- n2 <- power$n[i]
predictorData <- data.frame(Diet = rep(c("c","hf"),c(n1,n2)) %>% as.factor)
simResults <- t(sapply(1:nSim,simLm,form = ~Diet,data = predictorData,betas = c(b0,b1),sd = sd,contrasts = "Diethf = 0"))
power$power[i] <- mean(simResults[,"pvalues"] < alpha)
}
power
```
```{r}
power %>%
ggplot(aes(x=n,y=power)) +
geom_line()
```
#### Bestudeer power in functie van steekproefgrootte en effectgrootte
Merk op dat
- het teken van delta arbitrair is omdat we tweezijdig testen.
- het intercept arbitrair is omdat we alleen testen op $\beta_1$
- We nemen daarom typisch $\beta_0 = 0$
```{r}
nSim <- 1000
b0 <- 0
deltas <- c(1,2,3,5,10)
sd <- round(miceSum$sd[2],1)
ns <- c(3,5,10,20,25,50,75,100)
power <- data.frame(b1=rep(deltas,each=length(ns)),
n=rep(ns,length(deltas)),
power=NA)
for (i in 1:nrow(power))
{
b1 <-power$b1[i]
n1 <- n2 <- power$n[i]
predictorData <- data.frame(Diet = rep(c("c","hf"),c(n1,n2)) %>% as.factor)
simResults <- t(sapply(1:nSim,simLm,form = ~Diet,data = predictorData,betas = c(b0,b1),sd = sd,contrasts = "Diethf = 0"))
power$power[i] <- mean(simResults[,"pvalues"] < alpha)
}
```
```{r}
power %>%
ggplot(aes(x=n,y=power,col=b1%>%as.factor)) +
geom_line()
```
Merk op dat de power curves wat schokkerig zijn omdat we maar een beperkt aantal steekproefgroottes en een beperkt aantal simulaties uitvoerden.
Voor de lage powers is het aantal simulaties zeker onvoldoende om heel betrouwbare schattingen van de power te bekomen.
# Opmerkingen
- De code kan eenvoudig aangepast worden naar andere designs (zie oefeningen)
- predictor data
- formula
- contrast
- De simulaties kunnen lang duren wanneer je veel scenario's evalueert.
- Meer efficiënte code met matrices
- Voor two group comparison bestaat een analytische oplossing.
## Meer efficiënte code met matrices
Code is veel sneller. We simulaleren nu 20 keer meer experimenten in een veel kortere tijd!
```{r}
nSim <- 20000
b0 <- 0
sd <- round(miceSum$sd[2],1)
ns <- c(3,5,10,20,25,50,75,100)
deltas <- c(1,2,3,5,10)
powerFast <- matrix(NA,nrow=length(ns)*length(deltas),ncol=3) %>% as.data.frame
names(powerFast) <- c("b1","n","power")
i <- 0
for (n in ns)
{
n1 <- n2 <- n
### Simulation
predictorData <- data.frame(Diet = rep(c("c","hf"),c(n1,n2)) %>% as.factor)
design <- model.matrix(~Diet,predictorData)
L <- limma::makeContrasts("Diethf",levels = colnames(design))
for (b1 in deltas)
{
ySim <- rnorm(nrow(predictorData)*nSim,sd=sd)
dim(ySim) <-c(nrow(predictorData),nSim)
ySim <- ySim + c(design %*%c(b0,b1))
ySim <- t(ySim)
### Fitting
fitAll <- limma::lmFit(ySim,design)
### Inference
varUnscaled <- c(t(L)%*%fitAll$cov.coefficients%*%L)
contrasts <- fitAll$coefficients %*%L
seContrasts <- varUnscaled^.5*fitAll$sigma
tstats <- contrasts/seContrasts
pvals <- pt(abs(tstats),fitAll$df.residual,lower.tail = FALSE)*2
i <- i+1
powerFast[i,] <- c(b1,n,mean(pvals < alpha))
}
}
```
```{r}
powerFast %>%
ggplot(aes(x=n,y=power,col=b1%>%as.factor)) +
geom_line()
```
## Meer efficiënte code gebaseerd op de analytische oplossing voor een two group comparison
- Voor de two sample t-test bestaat er een analytische oplossing om de power te berekenen.
- In deze context maakt men typisch gebruik van de "Cohen's effect size":
$D = \frac{\delta}{SD}$
```{r}
power.t.test(n=8, delta = delta, sd = sd, type='two.sample')
```
Merk op dat de power overeenkomt met degene die we via simulaties hebben geschat!
```{r}
b0 <- 0
sd <- round(miceSum$sd[2],1)
ns <- c(3,5,10,20,25,50,75,100)
deltas <- c(1,2,3,5,10)
powerTheo <- data.frame(deltas=rep(deltas,each=length(ns)),
n=rep(ns,length(deltas)),
power=NA)
powerTheo$power <- apply(powerTheo[,1:2],1,function(x) power.t.test(delta=x[1],n=x[2],sd=sd,type="two.sample")$power)
```
```{r}
powerTheo %>%
ggplot(aes(x=n,y=power,col=deltas%>%as.factor)) +
geom_line()
```